2. 江苏省环境监测中心,江苏 南京 210019
2. Jiangsu Environmental Monitoring Center, Nanjing, Jiangsu 210019, China
生物多样性与人类的生活、生产和生态环境息息相关,是人类赖以生存和发展的重要基础,在维持生态平衡、环境稳定等方面发挥着极其重要的作用[1-4]。昆虫作为生物多样性的组成成分,在维持生态系统功能的过程中发挥了关键作用[5]。鉴于昆虫体型小,生活周期短,对不断发生的细微环境敏感并能做出快速反应等特点,生态系统中昆虫多样性的动态变化对于指示整个系统的变化程度具有举足轻重的地位和作用[6]。但传统的昆虫形态学鉴定难度大,需要很强的专业性和标本的完整性[7-8],且科级水平以下的鉴定特征模糊甚至缺乏,往往难以准确鉴定到属和种,有可能降低评价等级的准确性和精确性,因此亟需新的调查方法满足现阶段日益增长的对陆生昆虫生物多样性评估的需求[9]。
环境DNA(Environmental DNA),即直接从环境样本中获取的不同生物基因组DNA的复杂混合物,来源于生物体脱落的细胞、排泄物等,最长可在环境介质中保存数十万年[10-13]。环境DNA宏条形码技术是指通过对从环境样本中提取到的DNA进行聚合酶链式反应(PCR)扩增和高通量测序来完成物种鉴定的技术[14]。环境DNA宏条形码技术与其他监测方式相比,具有更高的分辨率、更小的物种扩增偏差和更优异的检测能力[15]。目前,环境DNA技术已用于评估植物[16]、真菌[17]和鱼类[12]群落,监测入侵物种,并在稀有种、隐存种的检测鉴定中表现出优势[18]。马氏网是陆生昆虫生物多样性调查的主要方法之一,本研究探索目标为环境DNA宏条形码技术应用于马氏网昆虫鉴定是否能提升其速度以及准确性,从而为之后的陆生昆虫多样性调查提供新思路。
如何科学评估与人类活动相关的不同林地类型的陆生昆虫多样性,一直是森林生物多样性保护成效评估的重要内容之一。本研究通过应用环境DNA宏条形码技术检测人工林、杂木林和天然次生林地的昆虫多样性,目标一是评估利用环境DNA宏条形码技术检测马氏网内昆虫标本的可行性;二是验证林地昆虫多样性丰富程度与受人类活动干扰的强度相关假说,即自然天然次生林>杂木林>人工林[19]。
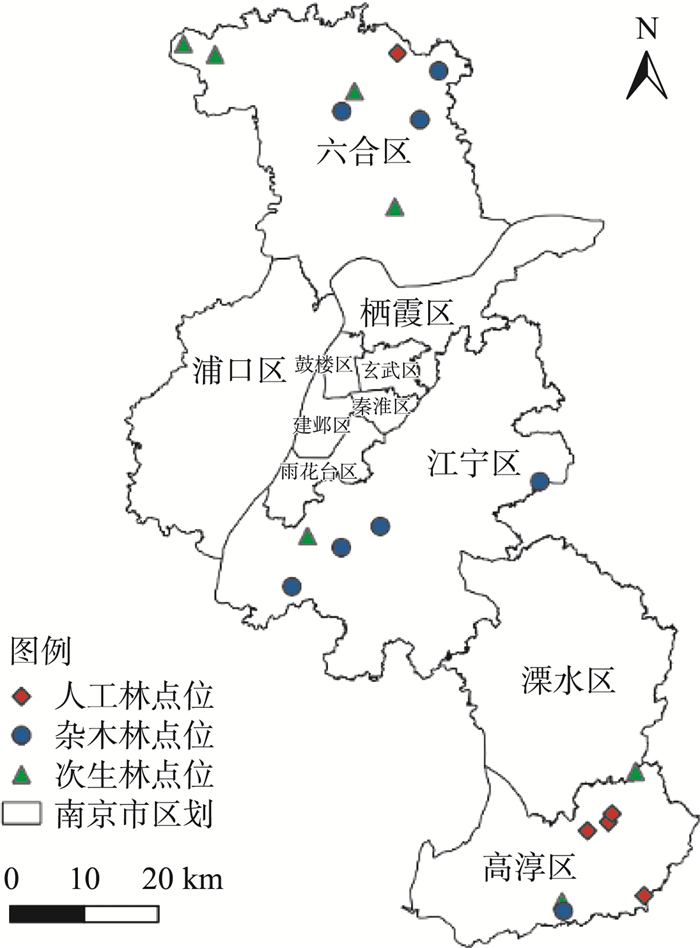
1 材料与方法 1.1 研究区域和样点设置南京市处于东洋古北的中间过渡地带,植被结构、地形地貌多样,为陆生昆虫的繁殖和扩散提供了良好条件。本研究选择人工林(人为栽培的马尾松、杨树林等),杂木林(砍伐后自然次生的土著落叶阔叶树为主的林地)和天然次生林(以阔叶林为主,乔木和灌木混生)作为调查林地。共设置20个样点(编号S01—S20),其中人工林地5块,杂木林地8块,天然次生林7块。采样点位置信息和分布示意见表 1和图 1。
| 表 1 采样点位置信息 |
|
图 1 采样点位分布示意 |
每块林地选中合适的地点架设1个马氏网,在马氏网的收集容器内装入不少于容器2/3体积的无水乙醇。调查采集时间为2022年3月18—2023年5月29日,取样瓶每2周更换一次[19],共采集7次,140个样本,总计184条样线,310个样方。采集到的标本均使用酒精浸泡保存。
1.3 DNA提取与扩增每份样品到达实验室后,首先排干收集液保留昆虫,加入新鲜的无水乙醇500 mL后在室温下存储放置5个月[18]。5个月后,采用无菌的筛网(孔径为0.5 mm)分离乙醇保存液和昆虫标本,随后用孔径为0.45 μm的玻璃微纤维滤膜(GF/F,沃特曼公司)过滤保存液。滤膜转移入5 mL无菌冻存管中,置于-80 ℃的冰箱中保存。实验所用的材料和设备都需要严格的消毒或灭菌处理。为了监测实验过程中可能出现的污染,每组样品处理完毕后,过滤500 mL的无水乙醇溶液作为阴性对照。
将20个样点分2组进行环境DNA的提取和PCR扩增,每组包括10个样点收集的样品以及1个阴性对照。环境DNA扩增前先用无菌剪刀将滤膜剪成3 mm2的碎片,使用Ezup柱式动物基因组DNA抽提试剂盒[生工生物工程(上海)股份有限公司]从滤膜中提取环境DNA。
实验中采用了正向引物mlCOlintF:5′-GGWACWGGWTGAACWGTWTAYCCYCC-3′,反向引物jgHCO219:5′-TANACYTCNGGRTGNCCRA ARAAYCA-3′,两者都在5′-末端添加了8个核苷酸长度的标签,以便在测序中识别不同样本。目标DNA片段的预期长度约为313碱基对(bp)[20-21]。
PCR反应总体系为25 μL,95 ℃预变性3 min,95 ℃变性30 s、循环30次,50 ℃退火30 s,72 ℃延伸30 s,72 ℃终延伸5 min。PCR产物于4 ℃冷藏保存。
1.4 分析方法 1.4.1 生物信息学分析利用SEED 2软件(用于分析高通量扩增子测序数据的平台)对高通量测序结果进行序列分析,通过使用“Blastn”(用于比较核苷酸序列的生物信息学工具)和“Identification”(物种鉴定)功能对这些操作分类单元(OTUs)序列进行比对和物种注释,从而获得环境样品中存在的物种信息。具体的操作步骤如下:对原始数据中的“Clean data”进行解压,并将“Paired-end reads”(从一个DNA片段的两端分别进行短序列的测序,生成2个互补的读段)中的双端序列合并;筛选出质量分数(Quality scores,Q)≥30的序列;根据上游和下游引物对应的标签组合进行序列检索,然后删除标签和引物序列;通过选择“Chimera-clean”(在生物样本的高通量测序后,从测序数据中识别并去除嵌合体序列)功能,使用Vsearch(用于处理和分析核苷酸序列数据的生物信息学工具)检测序列中的嵌合体,并保留非嵌合体序列。为了把环境DNA样本中的序列分类到相应的物种,基于一定的相似度标准,将序列聚类成OTUs;使用Vsearch工具,根据97%的相似度阈值来对序列进行OTUs聚类。在本研究中,使用了包括NCBI(美国国家生物技术信息中心)和GenBank数据库(NCBI维护的核苷酸序列数据库)在内的本地数据库进行序列比对,此过程通过“Blastn”功能实施。序列比对完成后,通过联网操作获取相关的物种分类信息。
在对环境DNA的物种注释结果进行筛选时,本研究首先移除了阴性对照以及样本中低丰度的序列(序列丰度 < 0.005%);随后,去除reads(短序列片段) < 10的OTUs,最终只保留相似度≥85%且覆盖度≥50%的序列。在此筛选基础上,进一步细化数据集,筛选出昆虫纲(Insecta)中属于陆生昆虫(除去蜉蝣目、毛翅目等典型的水生昆虫)的分类单元。在种、属、科级的分类水平上,相似度阈值分别设定为98%~100%(种级),90%~98%(属级),85%~90%(科级)。
1.4.2 统计分析研究使用香农-维纳(Shannon-Wiener)多样性指数、辛普森(Simpson)多样性指数[22]、Chao1指数[23]、ACE指数[24]评估昆虫生物多样性,均采用R语言中的vegan包(数据分析工具包)进行计算。
Shannon-Wiener多样性指数结合了物种丰富度和均匀度的信息,反映了群落的多样性水平。它考虑了物种的相对丰度,并且数值越高,表示群落的多样性越高。
Simpson指数度量了群落中任意2个个体被抽中属于同一物种的概率。它强调了物种的优势程度,值越小表示多样性越高。
Chao1指数主要用于估计群落中未被观察到的物种总数,特别适用于物种丰富度较高但个体数较少的样本。它基于稀有物种的数量来预测总物种数,有助于估计群落的真实物种丰富度。
ACE指数(Abundance-based Coverage Estim-ator) 是一种估计群落中总物种丰富度的方法,特别关注低丰度物种。它通过考虑少数几个个体被采集到的物种,来估计未被检测到的物种数量。
2 结果与分析 2.1 物种组成所有样品中共注释到昆虫纲595个OTUs合计711 624条序列。基于环境DNA宏条形码数据的昆虫群落结构见表 2。由表 2可见,舍弃毛翅目、蜉蝣目等典型的水生昆虫数据,在20个样点和2组阴性对照组中共注释到陆生昆虫11目101科330属465个OTUs 521 219条序列,阴性对照仅占总序列数的0.01%。双翅目的OTU丰度最高,其次是半翅目、鳞翅目。虽然鳞翅目在科级和种级水平都多于半翅目,但OTU丰度却小于半翅目。
| 表 2 基于环境DNA宏条形码数据的昆虫群落结构 |
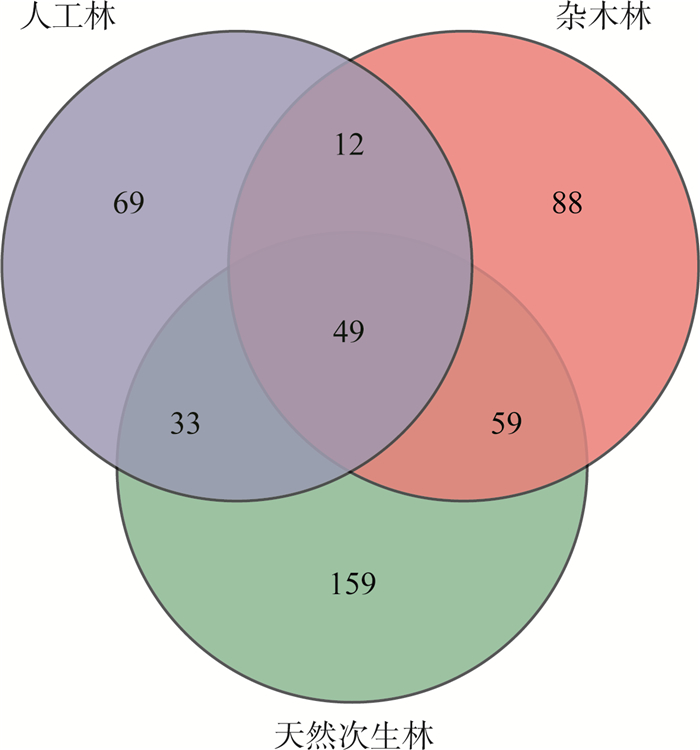
不同林地间昆虫OTU的维恩(Venn)图分析见图 2。由图 2可见,天然次生林中检测到的昆虫OTU数量最多(159),其次是杂木林(88),人工林的昆虫OUT数量最少(69)。天然次生林、杂木林和人工林之间共有的OTU数为49种,表明尽管这些生境类型不同,但存在一定程度的生物多样性重叠。这种重叠可能反映了这些生境在功能或进化上的某些共性。这些发现为理解不同生境的生物多样性特征提供了重要视角,有助于指导未来的环境保护和生态恢复策略。
|
图 2 不同林地间昆虫OTU的维恩图分析 |
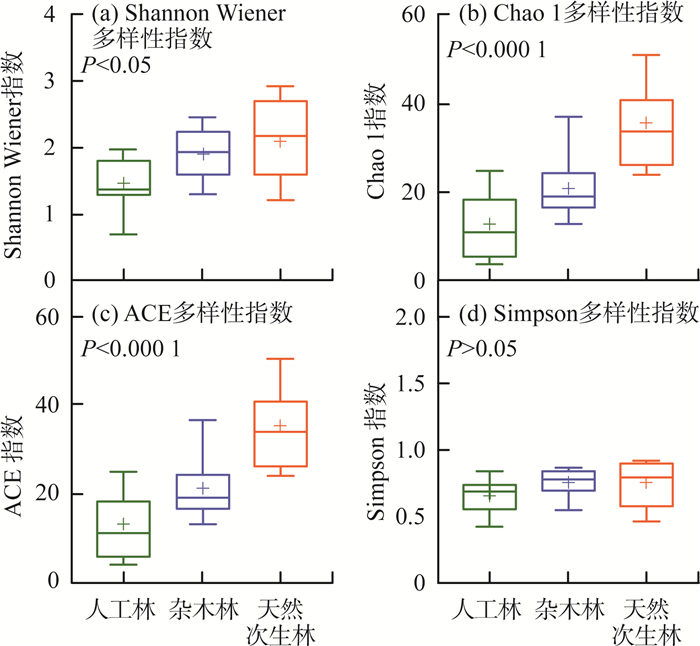
基于环境DNA宏条形码技术下不同生境的多样性指数见图 3(a)—(d)。由图 3可见,从多样性指数看,3种林地之间的Shannon-Wiener指数差异显著(P < 0.05),天然次生林的Shannon-Wiener指数最高(2.23),人工林最小(1.42)。Chao1指数和ACE指数在3种林地间也表现出显著差异(P < 0.001)。Chao1指数中天然次生林最高(34.5),人工林最低(11.9);ACE指数中天然次生林最高,(33.1)人工林最低(10.6)。但是不同林地之间的Simpson指数没有显著差异(P>0.05)。
|
图 3 基于环境DNA宏条形码技术下不同生境的多样性指数 |
本研究通过应用环境DNA宏条形码技术,有效评估了马氏网中的昆虫生物多样性,是对快速、高效、低成本、批量处理林间马氏网样本进行陆生昆虫生物多样性检测的一次成功探索。本次调查采集时间为2022年3—5月,正值春末夏初,气候条件适宜,昆虫种类和数量处于攀升阶段[25]。陆生昆虫共注释11目101科330种465个OTUs,其中鳞翅目、双翅目和半翅目占很大比例,为优势类群,与以往研究调查的马氏网昆虫优势类群相吻合,进一步表明将环境DNA宏条形码技术运用在马氏网陆生昆虫调查中具备可行性与合理性。
本研究也发现环境DNA检测技术存在3个主要问题:(1)不同的引物可能带来检测偏差,本研究采用的是通用引物,无法有效地扩增出全部物种(如鞘翅目)。(2)环境DNA来源的不确定性,环境DNA来源于活体、死亡个体、粪便等,导致引入样本来源的偏差,无法提供准确的存在物种和数量信息。(3)数据库的缺失,目前仍缺乏一个统一完整的环境DNA条形码数据库,大型数据库NCBI、BOLD(生命条形码数据系统)等都是研究者自行上传的,其中存在大量冗余与错误序列,影响了参考数据库的准确性[26]。
通过应用环境DNA宏条形码技术对马氏网中昆虫的多样性指数进行分析,揭示了不同林地下昆虫群落的生物多样性指数具有显著差异。本研究表明天然次生林的生物多样性高于杂木林及人工林。Chao1指数、ACE指数及Shannon-Wiener指数的变化趋势一致,均是次生林最高,杂木林其次,人工林最低,且差异显著,与历史研究相吻合。而Simpson指数并无显著差异,可能是由于马氏网所能采集到的昆虫群落较为有限且重复,导致群落结构相似;或是采集到的优势物种数量过多,数量稀少的物种被忽略。林间生物多样性不一致可能与人类林业活动对不同林分的干扰程度有关[27],人工林树种组成单一、结构层次简单[28],难以支撑多种昆虫共存;过度的人类干扰,昆虫食物资源减少,林内空间异质性降低,导致其昆虫生物多样性显著少于其他2种林分。而天然次生林及杂木林树种丰富、垂直结构较为复杂[29],为昆虫食物来源和隐蔽场所,提高了昆虫多样性。科学地规划和管理植被结构,降低植被人为干扰程度是保护城市森林昆虫多样性的关键。
本研究通过马氏网法采集的昆虫虽不能反映所有昆虫类群,但在一定程度上表明人为干扰是影响昆虫多样性的重要因素之一,并评估了不同林分下昆虫多样性的差异。研究结果也表明了环境DNA宏条形码技术在检测马氏网昆虫群落中具备可行性及合理性,且能够提高批量昆虫鉴定的效率及精确性。虽然这一技术在应用于陆生昆虫鉴定时仍面临DNA来源的复杂性导致序列注释困难,不同的引物可能带来检测偏差,环境样品中DNA普遍含量低、降解严重等问题,但通过优化实验流程和分析管道,并结合其他手段,该技术有望成为一种高效低耗的大规模陆生昆虫多样性调查新方法,尤其对于一些隐蔽性强、活动性高的物种。长期监测不同生境下昆虫多样性的时空变化,也将为生物多样性保护提供科学数据支撑。
| [1] |
王梦, 姚飞, 宁少华, 等. 北京小西山昆虫资源多样性与保护分析[J]. 安徽农业科学, 2024, 52(5): 132-136, 143. |
| [2] |
杨俊, 袁菲, 王文学, 等. 百望山森林公园昆虫多样性调查分析[J]. 林业科技, 2023, 48(5): 39-43. |
| [3] |
李岩锐, 王莉英, 王潇, 等. 青海省祁连县昆虫多样性分析[J]. 内蒙古林业科技, 2024, 50(1): 61-64. |
| [4] |
ZOU J Y, CADOTTE M W, BÄSSLER C, et al. Wood decomposition is increased by insect diversity, selection effects, and interactions between insects and microbes[J]. Ecology, 2023, 104(12): e4184. DOI:10.1002/ecy.4184 |
| [5] |
WANG M, LUO A, ZHOU Q, et al. Research progress on insect diversity[J]. Biodiversity Science, 2022, 30(10): 22454. DOI:10.17520/biods.2022454 |
| [6] |
王梦, 姚飞, 宁少华, 等. 北京小西山昆虫资源多样性与保护分析[J]. 安徽农业科学, 2024, 52(5): 132-136, 143. |
| [7] |
MORA C, TITTENSOR D P, ADL S, et al. How many species are there on earth and in the ocean[J]. Plos Biology, 2011, 9(8): e1001127. DOI:10.1371/journal.pbio.1001127 |
| [8] |
HOPKINS G W, FRECKLETON R P. Declines in the numbers of amateur and professional taxonomists: Implications for conservation[J]. Animal Conservation, 2006, 5(3): 245-249. |
| [9] |
王萌, 金小伟, 林晓龙, 等. 基于环境DNA-宏条形码技术的底栖动物监测及水质评价研究进展[J]. 生态学报, 2021, 41(18): 7440-7453. |
| [10] |
SKRBINSEK T, JELENCIC M, WAITS L, et al. Monitoring the effective population size of a brown bear(Ursus arctos) population using new single-sample approaches[J]. Molecular Ecology, 2012, 21(4): 862-875. DOI:10.1111/j.1365-294X.2011.05423.x |
| [11] |
SIGSGAARD E, NIELSEN I B, BACH S, et al. Population characteristics of a large whale shark aggregation inferred from seawater environmental DNA[J]. Nature Ecology & Evolution, 2016, 1(1): 1-4. |
| [12] |
THOMSEN P F, KIELGAST J, IVERSEN L L, et al. Monitoring endangered freshwater biodiversity using environmental DNA[J]. Molecular Ecology, 2012, 21(11): 2565-2573. |
| [13] |
WILLERSLEV E, DAVISON J, MOORA M, et al. Fifty thousand years of Arctic veg-etation and megafaunal diet[J]. Nature, 2014, 506: 47-51. |
| [14] |
BRADLEY J C, EMMETT J D, ANDREW G, et al. Biodiversity loss and its impact on humanity[J]. Nature, 2012, 486(7401): 59-67. |
| [15] |
MU Y, ZHANG J, YANG J, et al. Enhancing amphibian biomonitoring through eDNA metabarcoding[J]. Molecular Ecology Resources, 2024, 24(4): e13931. |
| [16] |
YOCCOZ N G, BRÅTHEN K A, Gielly L, et al. DNA from soil mirrors plant taxonomic and growth form diversity[J]. Molecular Ecology, 2012, 21(15): 3647-3655. |
| [17] |
O'BRIEN H E, PARRENT J L, JACKSON J A, et al. Fungal community analysis by large-scale sequencing of environmental samples[J]. Applied and Environmental Microbiology, 2005, 71(9): 5544-5550. |
| [18] |
SHOKRALLA S, SINGER G A C, HAJIBABAEI M. Direct PCR amplification and sequencing of specimens' DNA from preservative ethanol[J]. Biotechniques, 2010, 48(3): 233-234. |
| [19] |
唐楚飞, 葛成, 曹烨, 等. 城市森林不同林分类型的昆虫多样性: 以南京紫金山南麓为例[J]. 生物多样性, 2023, 31(2): 100-112. |
| [20] |
DEAGLE B E, JARMAN S N, COISSAC E, et al. DNA metabarcoding and the cytochrome c oxidase subunit Ⅰ marker: Not a perfect match[J]. Biology Letters, 2014, 10(9): 235-242. |
| [21] |
LERAY M, YANG J Y, MEYER C P, et al. A new versatile primer set targeting a short fragment of the mitochondrial COI region for metabarcoding metazoan diversity: Application for characterizing coral reef fish gut contents[J]. Frontiers in Zoology, 2013, 10: 34-47. |
| [22] |
马克平, 钱迎倩, 王晨. 生物多样性研究的现状与发展趋势[J]. 科技导报, 1995(1): 27-30. |
| [23] |
CHAO A. Nonparametric estimation of the number of classes in a population[J]. Scandinavian Journal of Statistics, 1984, 11(4): 265-270. |
| [24] |
CHAO A, YANG M C K. Stopping rules and estimation for recapture debugging with unequal failure rates[J]. Biometrika, 1993, 80(1): 193-201. |
| [25] |
刘珊, 杨政, 周琼. 南岳衡山春季昆虫多样性调查[C]//湖南省昆虫学会, 湖北省昆虫学会, 河南省昆虫学会. 华中昆虫研究(第6卷). 长沙: 中南大学出版社, 2010.
|
| [26] |
谷思雨, 陈凯, 金小伟, 等. 水生生物环境DNA监测技术的发展、应用与标准化[J]. 水生生物学报, 2024, 48(8): 1443-1458. |
| [27] |
刘欢欢, 李晨阳, 李梦莉, 等. 广西马尾松人工林与毗邻天然林昆虫群落组成及多样性研究[J]. 西南林业大学学报(自然科学版), 2024, 44(2): 144-154. |
| [28] |
MENG J H, LU Y C, ZENG J. Transformation of a degraded Pinus massoniana plantation into a mixed species irregular forest: Impacts on stand structure and growth in Southern China[J]. Forests, 2014, 5(12): 3199-3221. |
| [29] |
周晓果, 温远光, 朱宏光, 等. 大明山常绿阔叶林冠层垂直结构与林下植物更新[J]. 应用生态学报, 2017, 28(2): 367-374. |
 2024, Vol. 16
2024, Vol. 16 

